基因组重复区域组装的新算法
时间:2020-09-09 14:50来源:未知 作者:二九 点击: 次
作为任何基因组测序项目中不可或缺的一步,从头组装使用一组读取数据(1,2)从头开始重建基因组。准确和完整的装配是至关重要的,因为下游分析将受到低质量组件的严重影响(3)。作为应用最广泛的组装策略,基于de Bruijn图的算法(4-8)将序列读取分解为kmers,然后通过分解图并找到计算成本相对较低的欧拉路径来推断序列(9,10)。虽然基于de Bruijn图的方法具有固有的高计算效率,但是由于kmer的大小有限,复杂的重复将导致大量的碎片化。此外,当重复长度超过读取长度时,会增加组装的难度(12,13)。
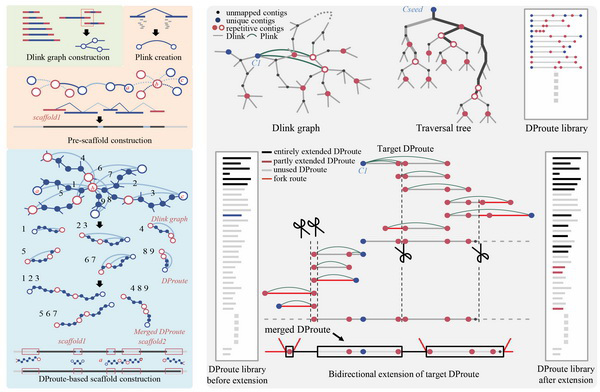
一个可能的解决方案是使用长读测序技术(即PacBio)来处理重复的基因组区域(14,15)。但是相对较高的测序成本和错误率限制了它们在从头基因组测序项目中的广泛应用(16,17)。另一种方法是利用Illumina测序产生的CONTIG之间的成对链接,然后执行脚手架过程,其中CONTIG将被排序、定向和连接。然而,基因组支架通常面临两个障碍,一个是来自作图偏差或错误的噪声配对链接,另一个是基因组中的重复区域。传统的scaffolding算法通常采用两种不同的策略(补充数据):(i)选择种子连接并从两端扩展到指定的最小重叠,且不匹配数量有限(18);(ii)以contigs为顶点,以成对链接为边,构建成对链接图,然后将图线性化(19–21)。这两种策略都是以后组装方式实现的,主要集中在消除成对链路噪声和扩展contigs上。例如,SSPACE(18)分层选择最长的contig,并通过一系列灵活的参数搜索适当的一致序列来扩展该contig。SCARPA(21)提出了两种去噪模型,即奇数周期横向模型和反馈弧集模型,对连续波进行定位和排序。SOPRA(22)采用了几种复杂的模型来消除成对链路的噪声,利用成对链路确定链路的方向,消除误装配。OPERA(23)通过逐步添加共享最可靠的成对连接的相邻连接来创建支架,并通过动态编程消除噪音。总的来说,这些基于成对链路的算法已经做出了大量的努力来建立标准来放弃冗余连接和减少成对链路噪声。然而,这些工具在很大程度上忽略了重复性接触。例如,Bambus2(24)、OPERA和SOPRA只是将这些重复的contigs屏蔽起来,以简化组装过程,而一些架子工可以组装一小部分与独特contigs紧密相关的contigs,但丢弃剩余的contigs。例如,如补充资料所示,重复性接触R4和R5因其前叉而被放弃,而R1、R2和R3可在支架末端成功连接,因为它们与非重复性contigs N1和N2之间共享成对连接。
成对连接作为一种间接连接,在本质上屏蔽了两个连接链之间的序列,使支架过程复杂化。此外,上述基于成对链接的方法绕过了嵌入在原始de Bruijn图中的直接链接,该链接是在将装配图分解为contigs的阶段拼接叉生成的。显然,合并直接链接将大大增加脚手架图的连通性,并简化contigs的布局。这样的优势将提高去噪的效率,更重要的是便于重复序列的组装。最近,SPAdes(25)已经被发展用来组装单细胞和多细胞细菌数据集,其中它使用成对组装图进行基因组组装。ExSPAnder(26)是一个由黑桃组成的模块,它在装配图上创建配对信息,并沿着高分边缘延伸支架。尽管SPAdes是为单细胞测序而设计的,但它在分离细菌基因组或微型异基因组上产生的组装长度和精确度都比其他汇编程序好(25)。然而,它仍然不能解决复杂区域,包括大量的短重复连接,部分原因是这些区域缺乏读对支持,并且表现出严重的映射偏差。考虑到短重复序列代表了大多数组装的contigs,这种情况甚至从根本上说是倾斜的。尽管大多数汇编器,如SOAPdenovo2(8)和ALLPATHS(4)已经将de Bruijn图中的原始连接信息合并到脚手架过程中,但是组装重复区域的挑战仍然没有得到解决。
在这里,我们提出了一个新的算法,inGAP-sf,基于直接链接图和成对链接图的结合来解决上述脚手架障碍。inGAP-sf采用直接链路来提供额外的路由,并降低了重复contigs富集区的复杂性。在成对链路的监督下,确定真正的路由,并通过分配给相应的路由对短重复链路进行分组。此外,我们建立了一个基于读对支持度估计的综合模型来消除噪声路由。通过对模拟数据集和真实数据集的测试,我们证明了inGAP-sf可以显著提高组装的连续性和基因组覆盖率,以及基因组的精确性。
